Informations générales
Description
L'objectif est de se familiariser avec les métadonnées etsoumettre des données et des métadonnées sur ENA (EBI)
Formateurs
Hélène Chiapello ;
Thomas Denecker ;
Jean-François Dufayard ;
Gautier Sarah ;
Frédéric de Lamotte ;
Paulette Lieby ;
Durée
1h
Progression dans le module 3
Introduction
Dans ce TP, nous allons réaliser les différentes étapes entre la collecte des données et la soumission dans une banque internationnale. Nous vous proposons un jeu de données de séquences pour ce TP d’un génome bactérien de S. salivarius (Télécharger l’archive);
Vous trouverez dans le dossier une fois décompressé, deux fichiers .fastq.gz.
Important : Ces données sont des données fictives extraites de METAGENOTE. Elles ne sont en rien significatives.
Comme ces données sont des données de séquences, nous allons les soumettre sur l’ENA, la banque européenne de séquences.
Partie 1 : Exploration de l’European National Archive (ENA)

L’European National Archive est une plateforme ouverte et soutenue pour la gestion, le partage, l’intégration, l’archivage et la diffusion de données de séquences. Pour en savoir plus.
Connection à l’ENA
Lors d’un dépôt de données sur l’ENA, il est nécessaire de créer un compte. Lors de ce TP, vous aurez besoin d’un compte personnel si vous souhaitez essayer de soumettre des données TEST. Vous pouvez cliquer ici pour vous connecter à l’instance de test de l’ENA.
Comment sont gérées les métadonnées sur l’ENA ?
Chaque base de données organise ses données différement. Concernant l’ENA, vous trouverez ci-dessous son modèle d’organisation des données et métadonnées de l’ENA :
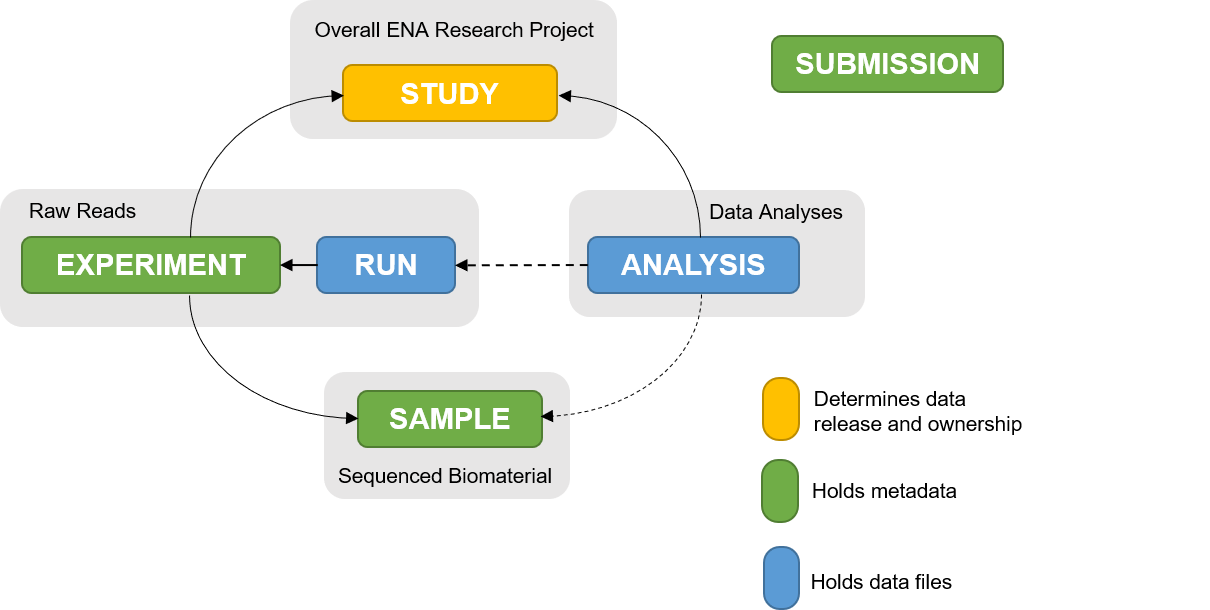
Extrait de la documentation de l'ENA
- Study: A study (project) groups together data submitted to the archive and controls its release date. A study accession is typically used when citing data submitted to ENA. Note that all associated data and other objects are made public when the study is released.
- Sample: A sample contains information about the sequenced source material. Samples are associated with checklists, which define the fields used to annotate the samples. Samples are always associated with a taxon.
- Experiment: An experiment contains information about a sequencing experiment including library and instrument details.
- Run: A run is part of an experiment and refers to data files containing sequence reads.
- Analysis: An analysis contains secondary analysis results derived from sequence reads (e.g. a genome assembly),
- Submission: A submission contains submission actions to be performed by the archive. A submission can add more objects to the archive, update already submitted objects or make objects publicly available.
Question 1
Quels sont le ou les objets de l’ENA qui sont associés à un vocabulaire contrôlé domaine dépendant appelé Checklist par l’ENA ?
StudySample
Experiment
Run
Analysis
Submission
Choisir une checklist correspondant au jeu de données
Une quantité minimale d’informations est requise lors de l’enregistrement des échantillons sur l’ENA et tous les échantillons doivent être conformes à une liste de contrôle (checklist) définissant des valeurs de métadonnées attendues. La liste de contrôle la plus appropriée pour l’enregistrement des échantillons dépend du type de l’échantillon. L’ensemble des checklists de l’ENA sont disponible ici. Pour le jeu de données de la bactérie S. salivarius, utiliser le mot-clé pathogen.
Question 2
Les checklists sont disponibles au format XML. Vous avez par exemple ci-dessous un extrait de la checklist ERC000028 :
<?xml version="1.0" encoding="UTF-8"?>
<CHECKLIST_SET>
<CHECKLIST accession="ERC000028" checklistType="Sample">
<IDENTIFIERS>
<PRIMARY_ID>ERC000028</PRIMARY_ID>
</IDENTIFIERS>
<DESCRIPTOR>
<LABEL>ENA prokaryotic pathogen minimal sample checklist</LABEL>
<NAME>ENA prokaryotic pathogen minimal sample checklist</NAME>
<DESCRIPTION>Minimum information required for a prokaryotic pathogen sample</DESCRIPTION>
<AUTHORITY>ENA</AUTHORITY>
<FIELD_GROUP restrictionType="Any number or none of the fields">
<NAME>Collection event information</NAME>
<FIELD>
<LABEL>isolation_source</LABEL>
<NAME>isolation_source</NAME>
<DESCRIPTION>describes the physical, environmental and/or local geographical source of the biological sample from which the sample was derived</DESCRIPTION>
<FIELD_TYPE>
<TEXT_FIELD/>
</FIELD_TYPE>
<MANDATORY>mandatory</MANDATORY>
<MULTIPLICITY>multiple</MULTIPLICITY>
</FIELD>
<FIELD>
<LABEL>lat_lon</LABEL>
<NAME>lat_lon</NAME>
<DESCRIPTION>geographical coordinates of the location where the specimen was collected</DESCRIPTION>
<FIELD_TYPE>
<TEXT_FIELD/>
</FIELD_TYPE>
<MANDATORY>recommended</MANDATORY>
<MULTIPLICITY>multiple</MULTIPLICITY>
</FIELD>
[...]
</FIELD_GROUP>
</DESCRIPTOR>
</CHECKLIST>
</CHECKLIST_SET>
Partie 2 - Utilisation de l’outil omicsBroker
Pour ce TP, nous allons utiliser omicsBroker, un outil de brokering développé par l’IFB. Il s’agit d’un prototype permettant d’annoter des données de séquences et de les soumettre à l’ENA.
Pour vous connecter à une instance de démonstration, cliquer sur le lien suivant : ici. Cette instance sera fermer dès la fin du TP. Si vous souhaitez refaire le TP, l’ensemble des étapes d’installation sont décrites sur GitHub.
- Attention 1 : omicsBroker est un un prototype qu’il ne faut pas utiliser avec des vraies données pour une vraie soumission !
- Attention 2 : Avez vous bien lu le attention 1 ?
Section Annotation du menu lattéral gauche
Cette section vous permet de créer un projet, de choisir une Checklist et remplir les métadonnées décrivant vos échantillons.
Cliquer sur le lien Project.
Consignes
Créer un projet en renseignant uniquement les champs obligatoires : Title, Alias et Description.
Cliquer sur le lien Samples
Choix de la checklist
Consignes
Sélectionner la checklist identifiée à la question précédente.
Rappel
Pour le jeu de données du génome bactérien de S. salivarius, la checklist est ERC000028
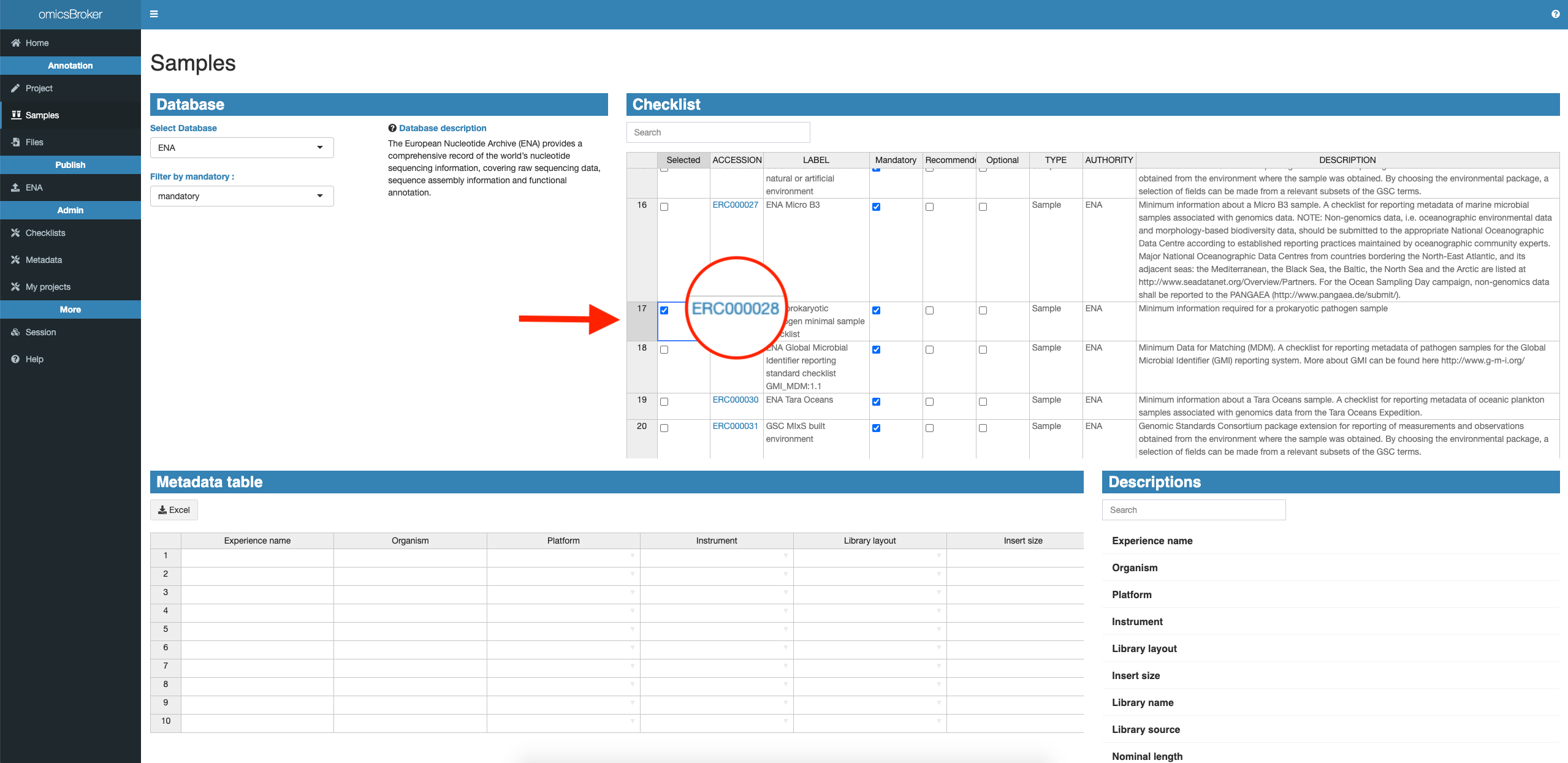
Le tableau dans la partie inférieure de la page Metadata table s’est mis à jour avec les champs correspondant à la Checklist sélectionnée.
Métadonnées
Consignes
Renseigner les champs obligatoires demandés en utilisant le contenu du DMP Opidor associé au jeu de données. Pour le trouver, aller sur Opidor - DMPs publics et rechercher le le plan IFB_Training_salivarius. Ensuite, télécharger le PDF qui contient toutes les informations nécessaires.
En cas de problème, le PDF peut être téléchargé ici ou ici.
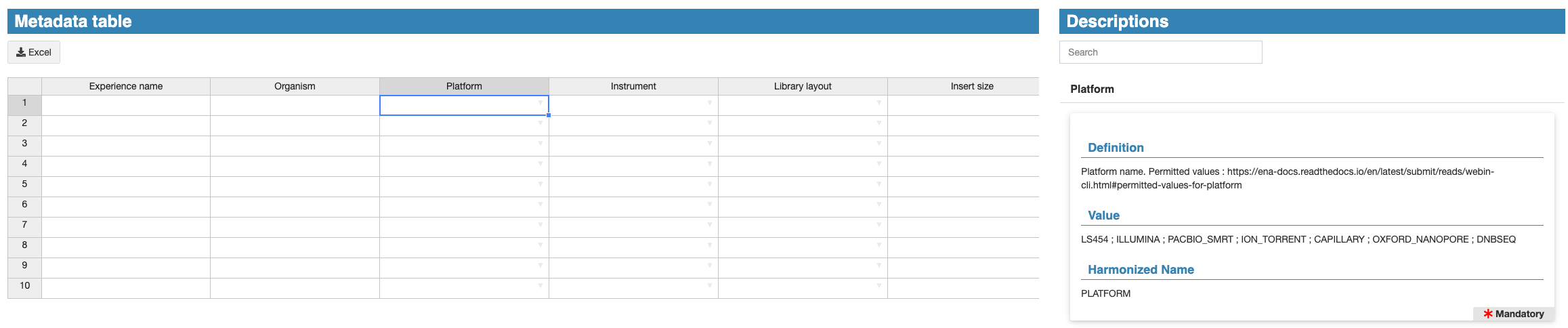
Pour savoir quel type d’information vous devez renseigner dans une cellule, cliquez sur la cellule en question et une notice vous donnera les informations nécessaires dans partie Descriptions (droite). Par exemple, ici les informations nécessaires dans la colonne Platform :
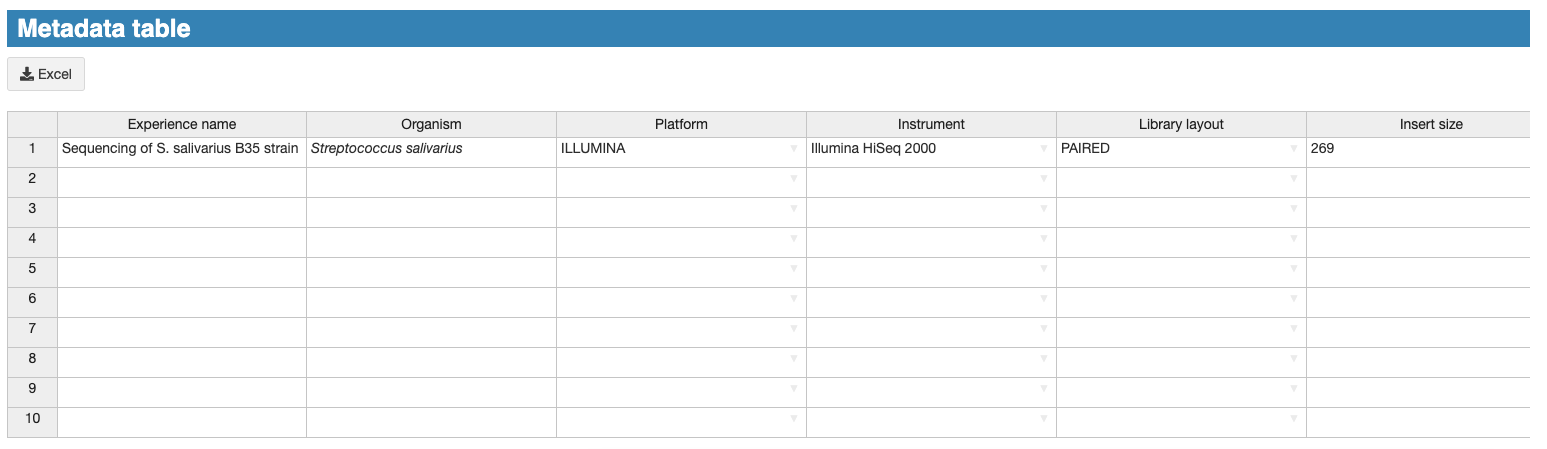
Ci dessous, un exemple d’une table remplie :
Cliquer sur le lien Files
Vous avez à votre disposition 2 fichiers une fois l’archive décompressées.
Rappel
Pour obtenir ces fichiers :
- Vous devez télécharger l’archive d’un génome bactérien de la bactérie S. Salivarius (Télécharger l’archive;
- Extraire les deux fichiers présents dans l’archive.
Consignes
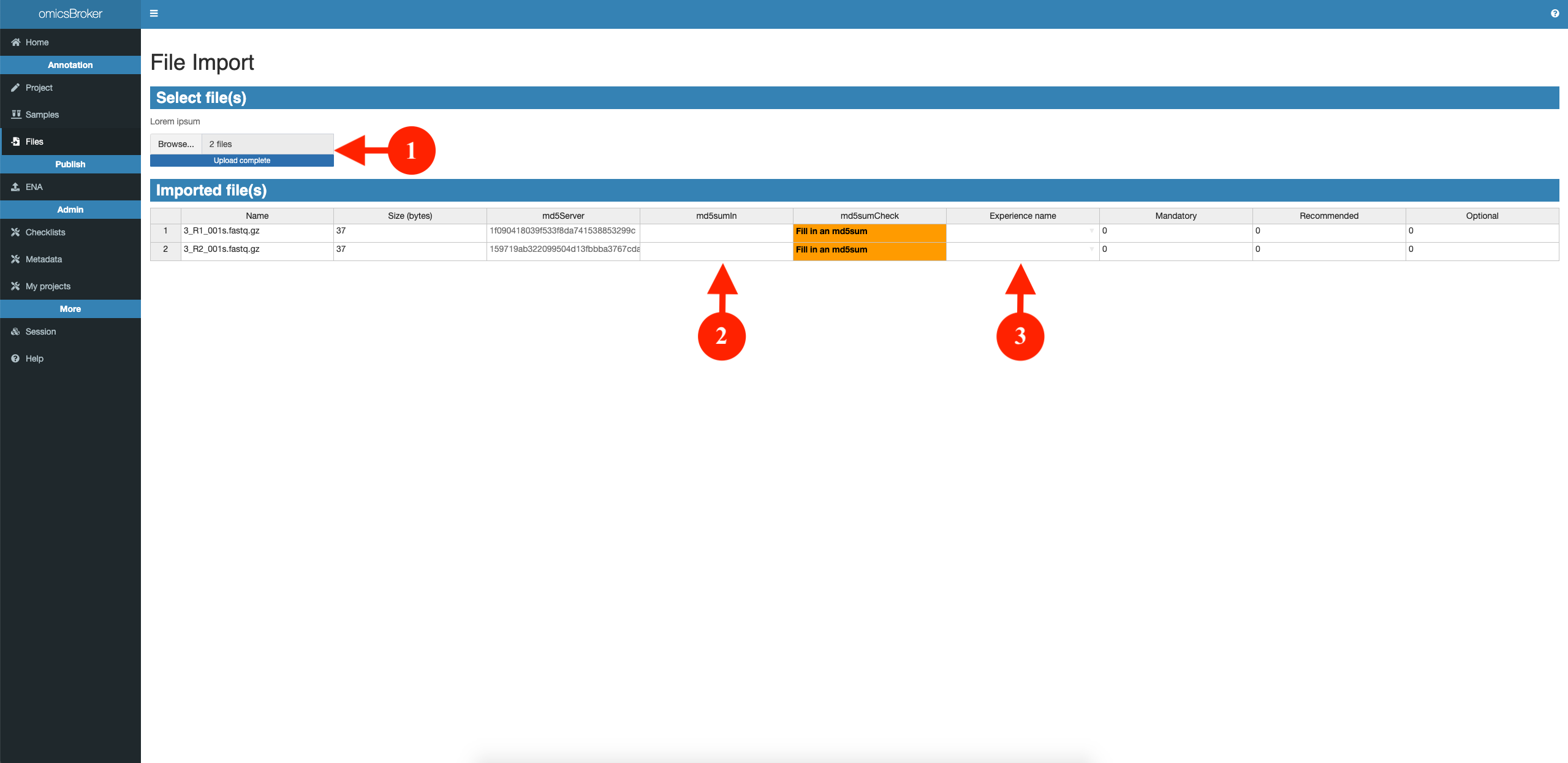
Téléverser ces deux fichiers dans omicsBroker en cliquant sur Browse puis en sélectionnant les fichiers (N°1 sur la figure ci-dessous). Vous pouvez aussi faire un `Drag & Drop.
Une fois téléversés, vous devriez avoir comme écran la figure suivante :
Consignes
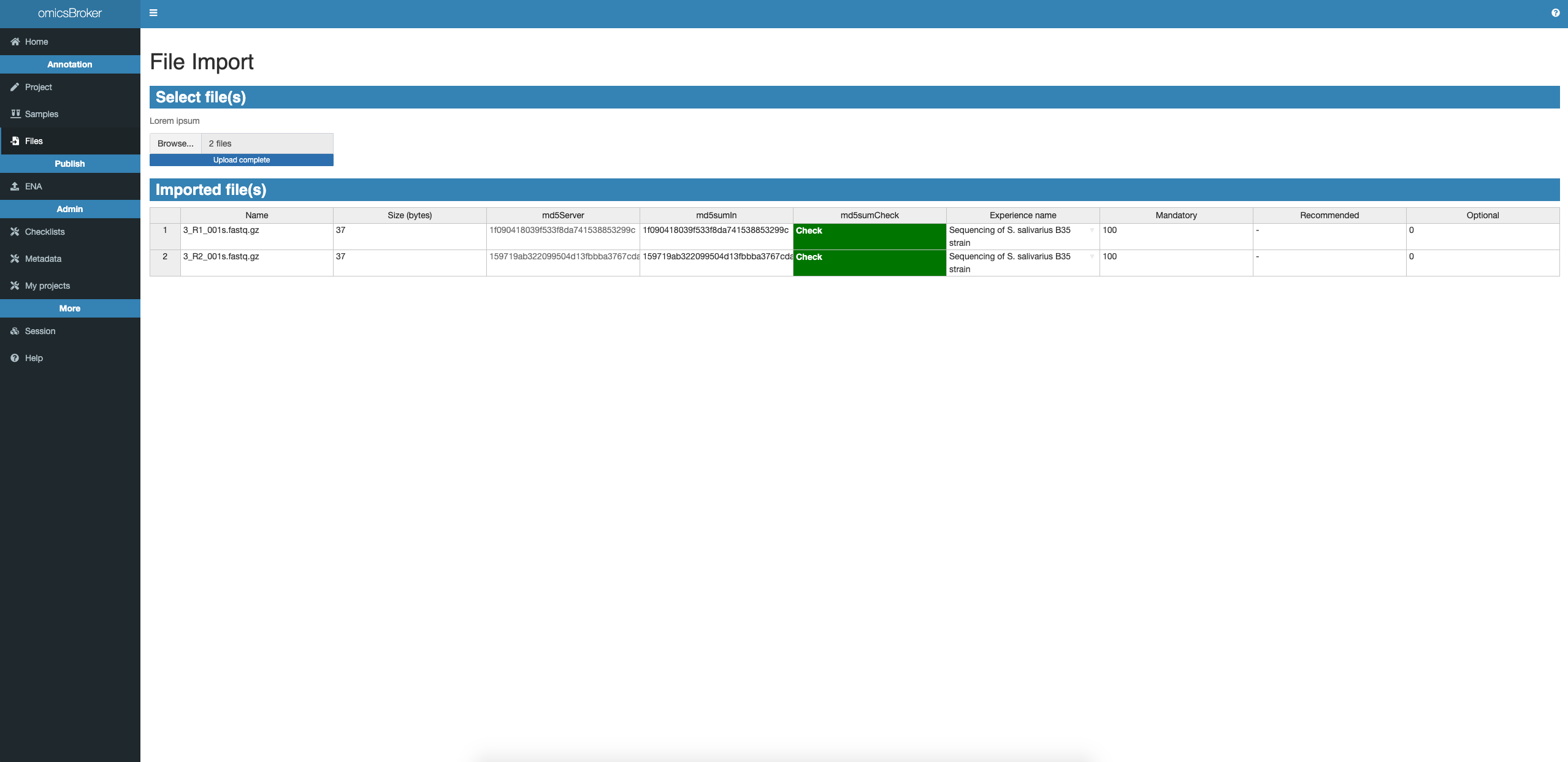
Renseigner le md5sum pour chaque fichier (N°2 sur la figure ci-dessus). Pour simplifier le TP et ne pas vous faire calculer en local ce md5sum, le tableau affiche le bom md5sum du fichier sur le serveur dans la colonne md5Server. Vous n’avez qu’à copier coller chaque valeur de cette colonne dans la colonne de droite md5SumIn.
L’objectif du md5sum est de s’assurer qu’aucune donnée n’a été perdue lors du téléversement. Pour obtenir le md5sum, nous vous conseillons la lecture suivante.
Consignes
Associer à chaque fichier un Experience name que vous avez renseigné dans la partie précédente (N°3 sur la figure ci-dessus). Pour cela, cliquer sur la cellule pour avoir la liste des Experience name disponibles dans la table metadata table et choisir le bon.
Une fois toutes ces informations renseignées, vous devez avoir quelque chose de similaire à la capture d’écran ci-dessous :
Section Publish du menu latéral gauche
Cette section permet de soumettre les fichiers de données et de métadonnées à l’instance de test de l’ENA.
Consignes
Cliquer sur l’onglet ENA dans la barre latérale.
Vous devez avoir à votre écran l’image suivante :
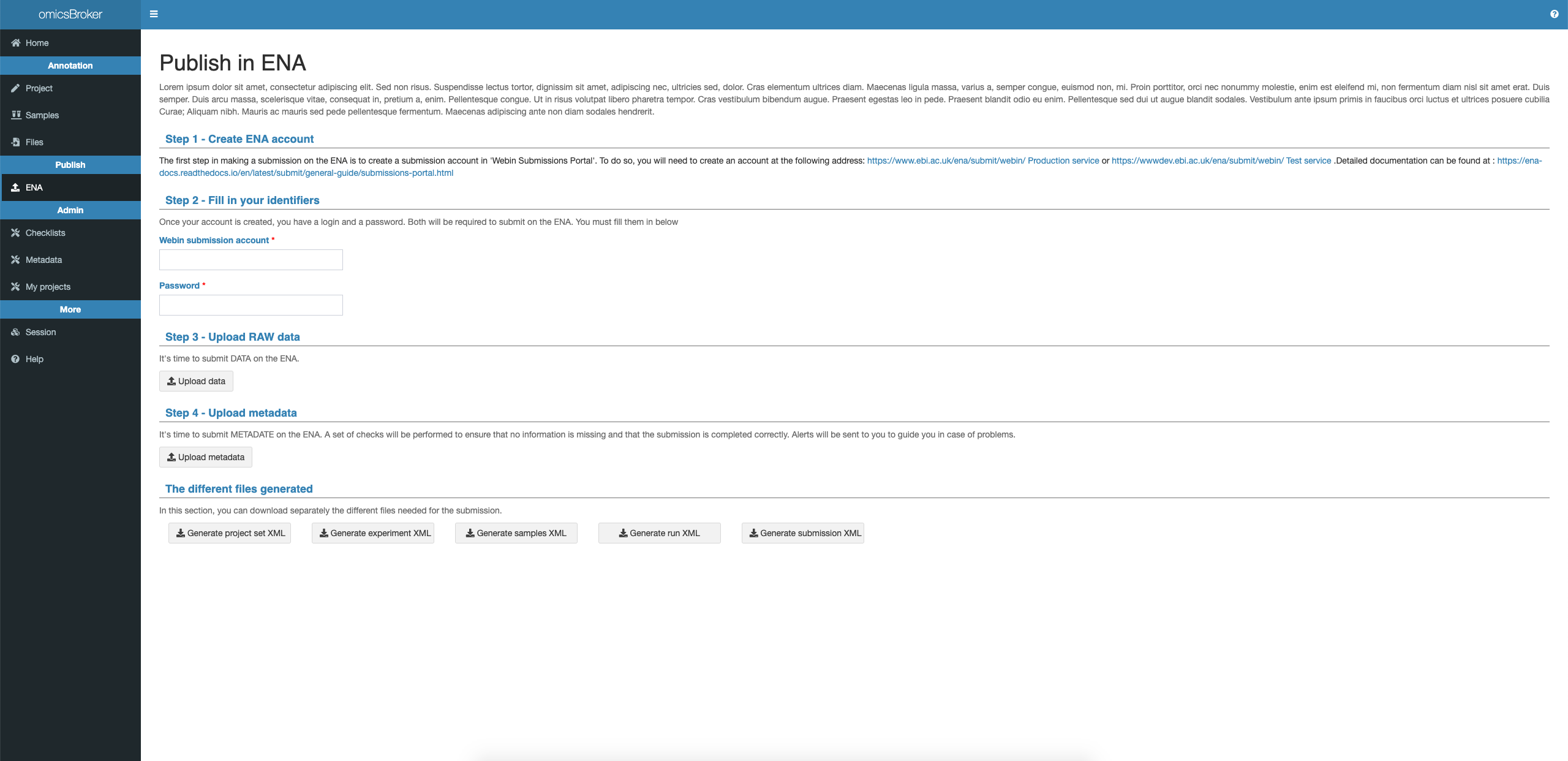
Consignes
Renseigner votre webin et votre mot de passe.
Par exemple :
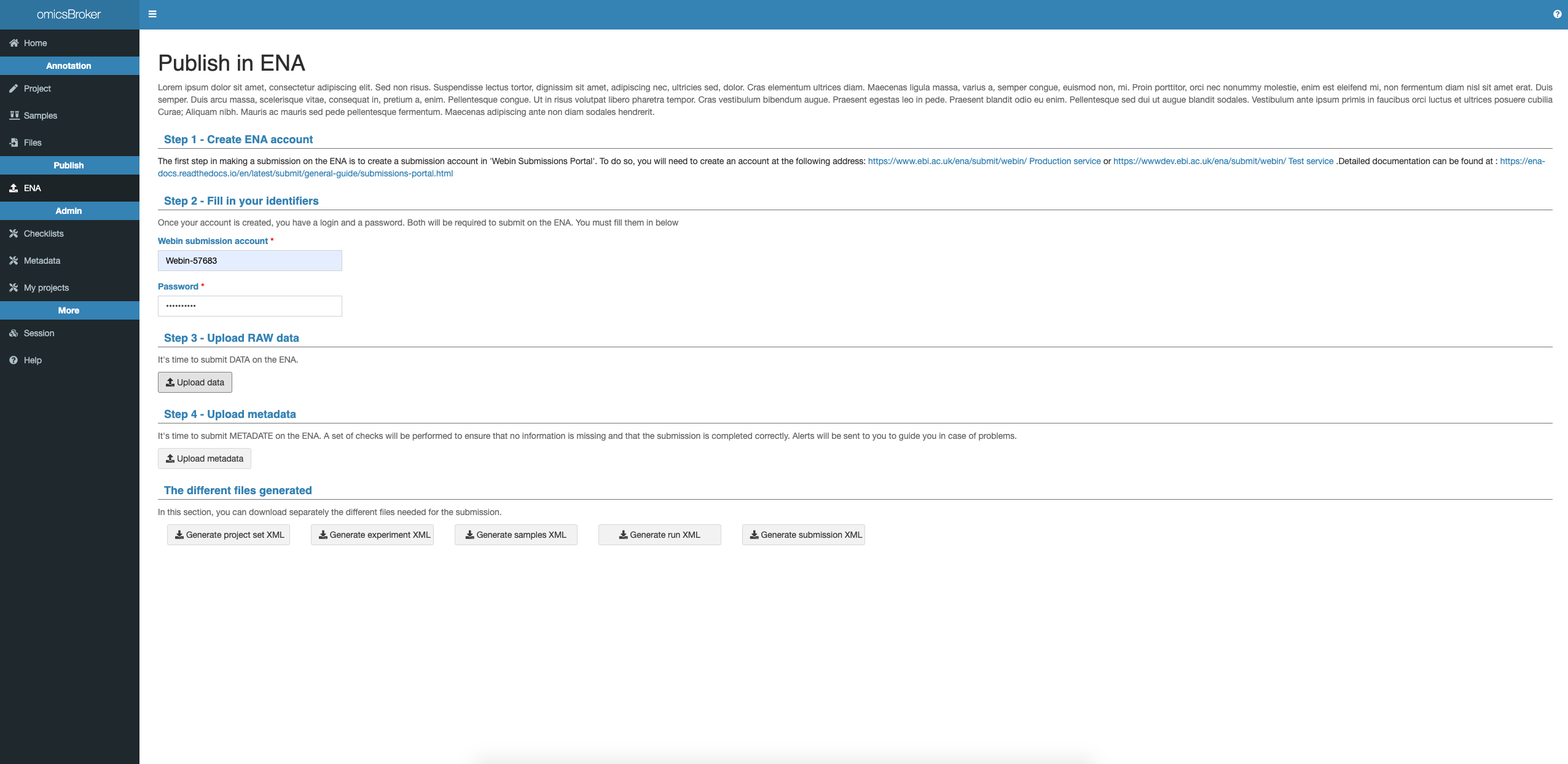
Envoyer les fichiers de données brutes sur l’instance de test de l’ENA
Consignes
Cliquer sur Upload data (Step 3).
En cas de succès, un message vous sera envoyé :
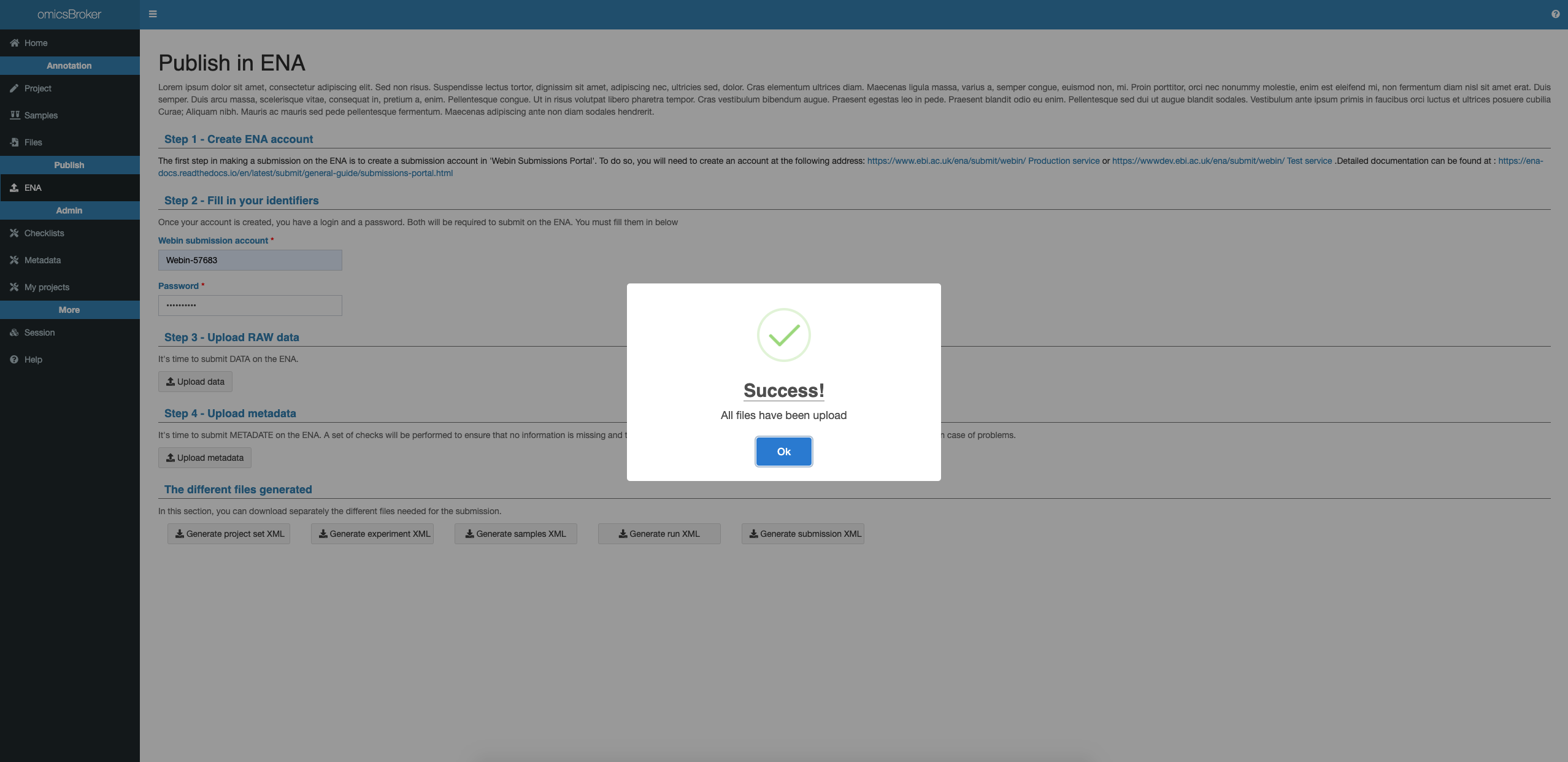
Note : si l’échec est lié au temps (message d’erreur renvoyé), recommencer jusqu’au succès.
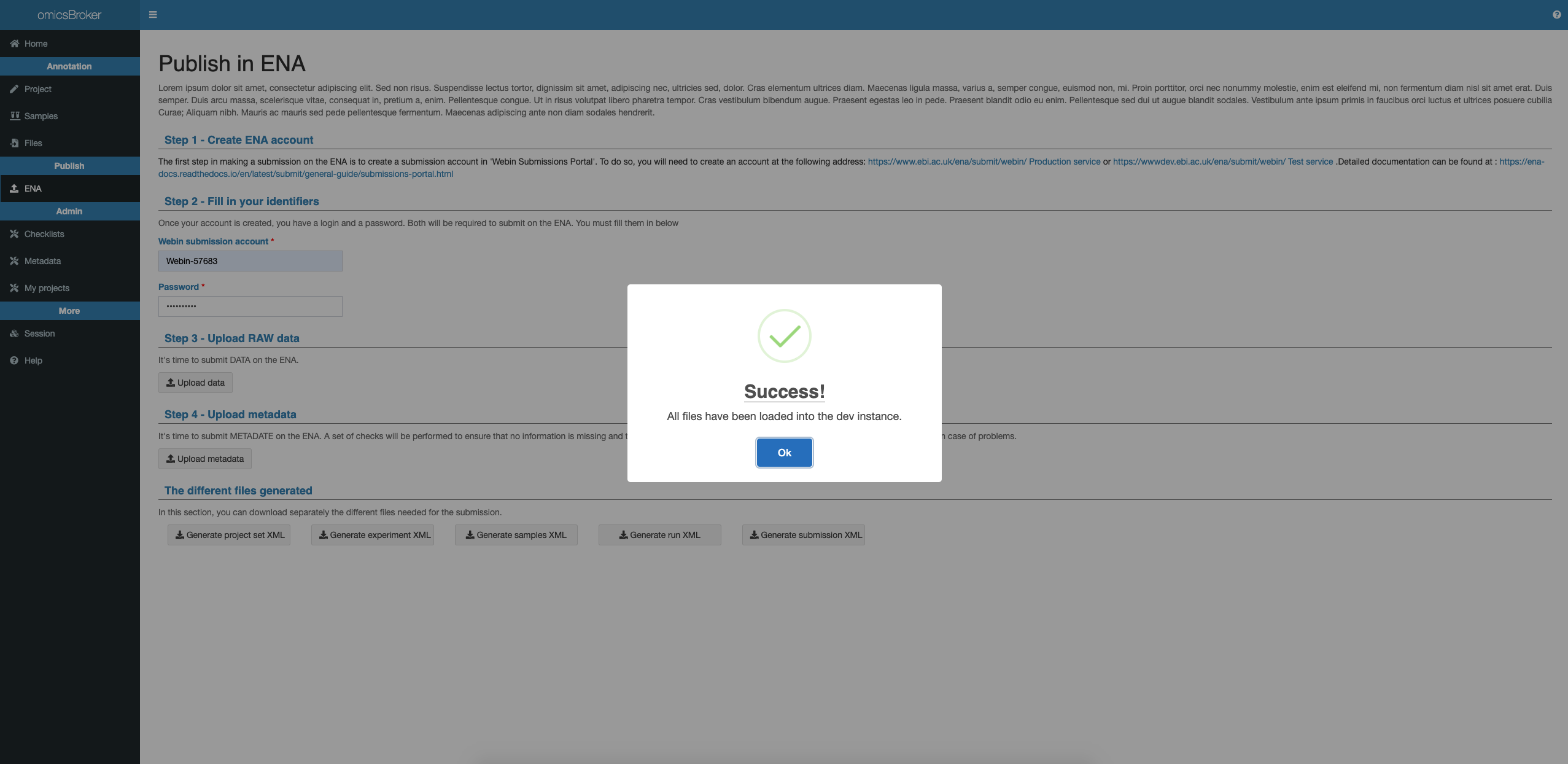
Envoyer les métadonnées sur l’instance de test de l’ENA
Consignes
Cliquer sur Upload metadata (Step 4).
En cas de succès, un message vous sera envoyé :
Décrire les fichiers XML de métadonnées générés
Consignes
Cliquer sur les différents fichiers XML générés par la soumission et commenter.
Exemple du fichier Project Set :
<PROJECT_SET>
<PROJECT alias="salivarius_dynamic">
<TITLE>Characterization of Streptococcus salivarius conjugatif elements content and diversity</TITLE>
<DESCRIPTION>Une description du projet</DESCRIPTION>
<SUBMISSION_PROJECT>
<SEQUENCING_PROJECT/>
</SUBMISSION_PROJECT>
</PROJECT>
</PROJECT_SET>
Exemple du fichier Experiment:
<EXPERIMENT_SET>
<EXPERIMENT alias="Sequencing_of_S__salivarius_B35_strain">
<TITLE>Sequencing of S. salivarius B35 strain</TITLE>
<STUDY_REF alias="XXXXXXXXXXXXXXXXXXX"/>
<DESIGN>
<DESIGN_DESCRIPTION/>
<SAMPLE_DESCRIPTOR accession="XXXXXXXXXXXXXXXXXXX"/>
<LIBRARY_DESCRIPTOR>
<LIBRARY_NAME/>
<LIBRARY_STRATEGY>WGS</LIBRARY_STRATEGY>
<LIBRARY_SOURCE>GENOMIC</LIBRARY_SOURCE>
<LIBRARY_SELECTION>RANDOM</LIBRARY_SELECTION>
<LIBRARY_LAYOUT>
<PAIRED NOMINAL_LENGTH="269"/>
</LIBRARY_LAYOUT>
<LIBRARY_CONSTRUCTION_PROTOCOL>Standart Illumina protocol</LIBRARY_CONSTRUCTION_PROTOCOL>
</LIBRARY_DESCRIPTOR>
</DESIGN>
<PLATFORM>
<ILLUMINA>
<INSTRUMENT_MODEL>Illumina HiSeq 2000</INSTRUMENT_MODEL>
</ILLUMINA>
</PLATFORM>
</EXPERIMENT>
</EXPERIMENT_SET>
Instance de test de l’ENA
Consignes
Vérifier que les différents fichiers de soumission sont bien sur l’instance de test de l’ENA. Pour cela, se connecter sur l’instance de test de l’ENA ici.
Une fois connecté, votre interface doit est comme ci-dessous :
En cliquant sur Studies Report :
En cliquant sur Samples Report :
En cliquant sur Runs Report :
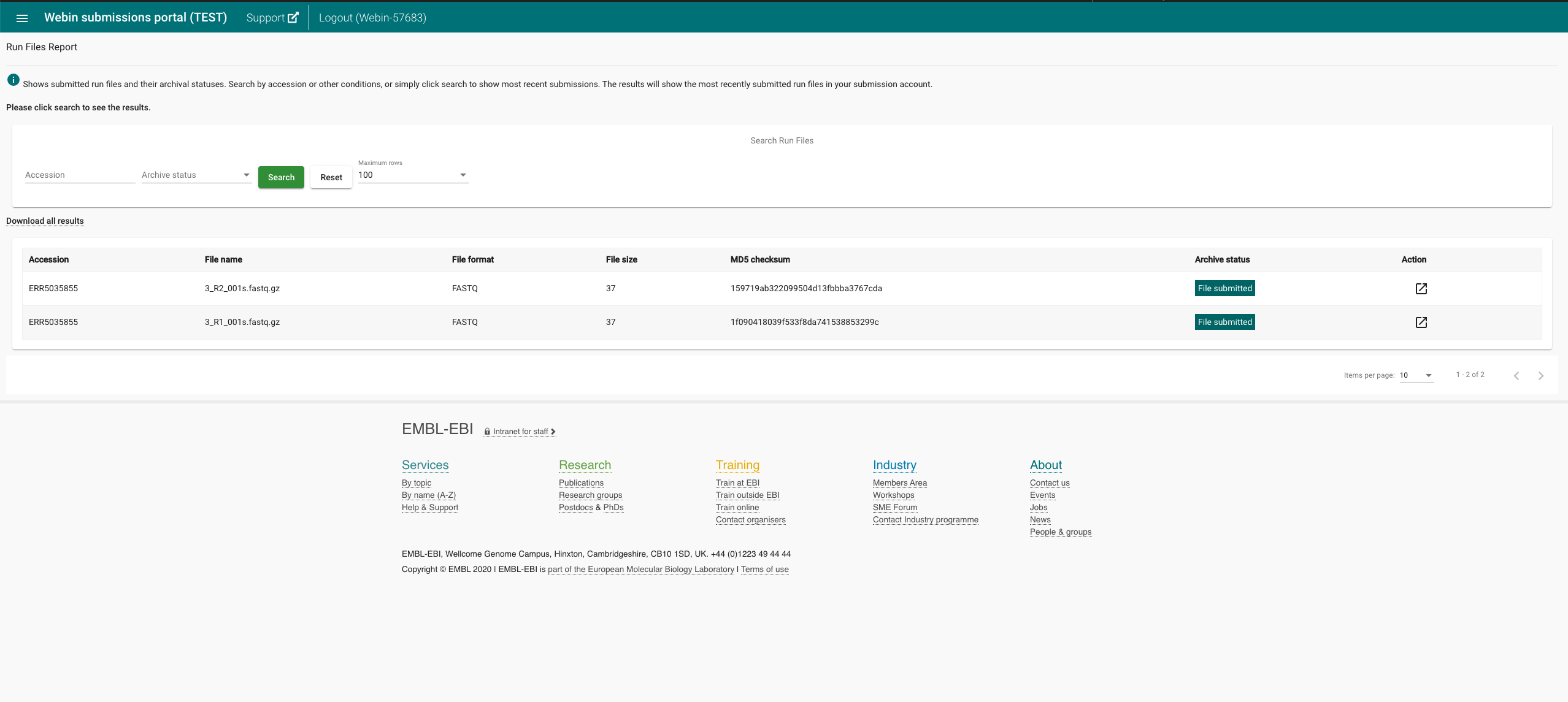
En cliquant sur Run Files Report :
Question 3
Quel est le numéro d’accession attribué à votre projet par l’ENA ?
Question Bonus : importation de métadonnées depuis le PGD
Via l’export du PGD en HTML, il est possible de pré-remplir certains items dans omicsBroker. Pour cela, vous devez :
- Exporter votre PGD au format HTML depuis Opidor. Vous pouvez cliquer ici puis enregistrer celui du PGD du TP.
- Importer le fichier HTML dans omicsBroker.
Consignes
Commenter les champs importés.