11ème école de bioinformatique AVIESAN - IFB - INSERM (EBAII 2022)
Initiation à l'utilisation du R markdown et knitR
Pourquoi utiliser ce type de fichier ?
Le fichier R markdown est un fichier dans lequel vous pouvez écrire du texte et des commandes R. Ce fichier permet ensuite de générer un rapport contenant à la fois les commandes et les résultats de ces commandes.
Commencer à utiliser ce fichier
Copier le fichier de départ R markdown dans votre espace projet, dans un dossier nommé Normalisation avec le Terminal de JupyterHub.
cd /shared/projects/<your_project>/
mkdir Normalisation
cp -r /shared/projects/form_2022_32/SingleCellRNASeq/ RMarkdown.header.Rmd /shared/projects/<your_project>/ Normalisation/
Dans Rstudio, ouvrir le fichier R markdown avec File > open File
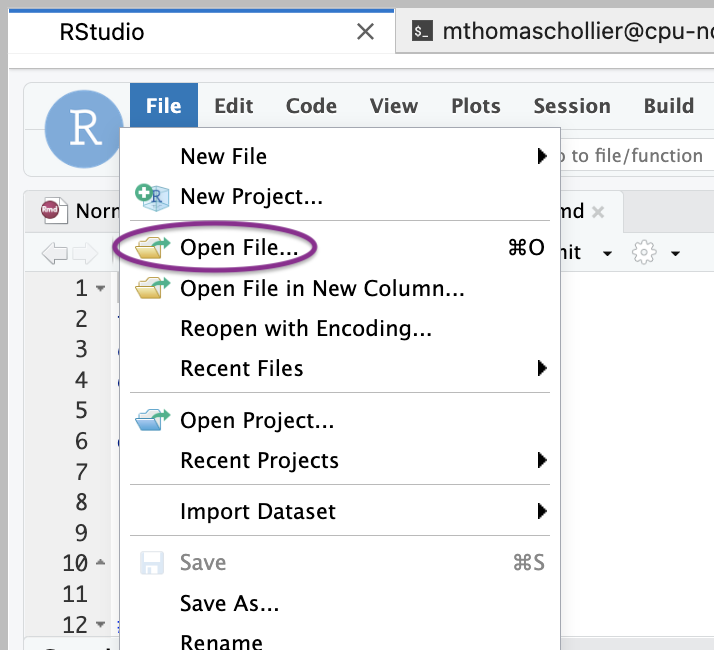
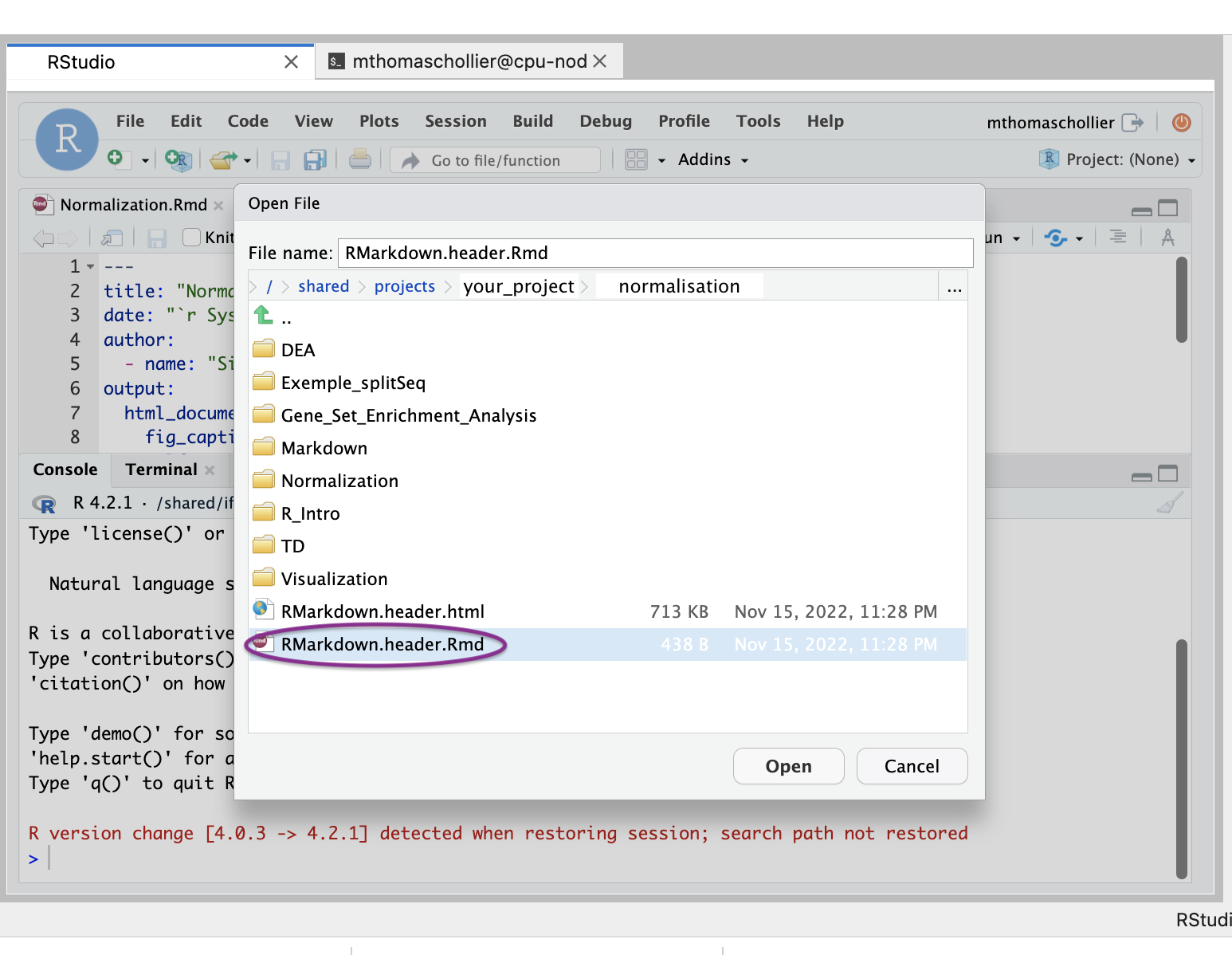
Choisir dans le dossier Normalisation le fichier nommé RMarkdown.header.Rmd
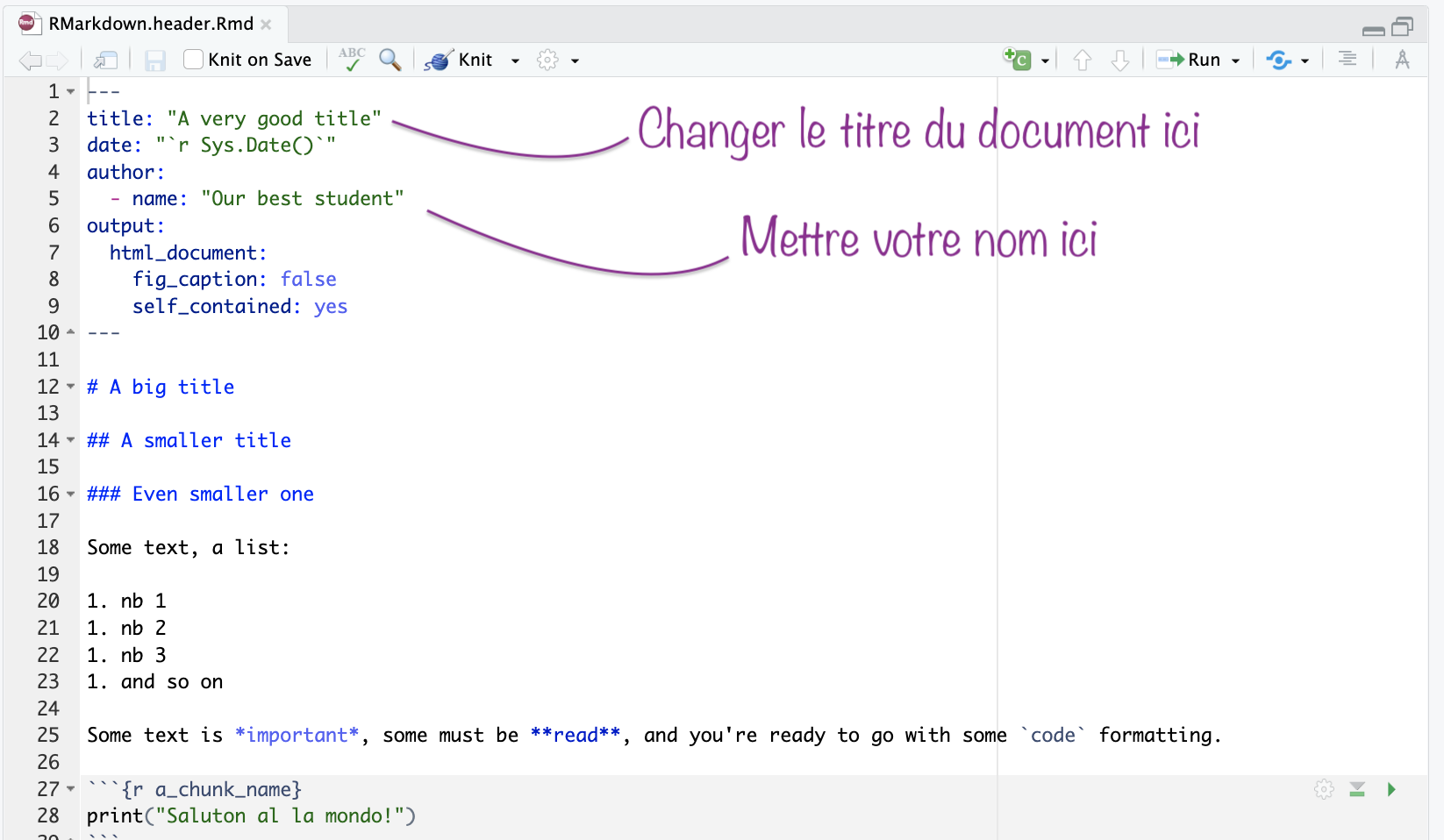
Le contenu du fichier s'affiche, et vous pouvez alors l'éditer pour y indiquer un titre et votre nom.
Ecrire du texte
Indiquer le texte de manière libre dans le fichier. Afin de rendre le texte plus lisible, vous pouvez le formatter :
- titres et sous-titres (avec des
#)
- listes (avec des
*)
- gras et italique (avec des
* ou des _)
Si vous voulez plus d'informations sur la syntaxe en R markdown, vous pouvez vous référer à cet aide mémoire.
Ecrire et lancer du code
Le code R s'écrit dans une section appelée "chunk" apparaissant sous forme grisée. Cette section commence et termine par 3 guillemets.
Nommer le chunk en remplacant a_chunk_name par un nom approprié pour cette étape. Ecrire une commande, ici print().

Pour lancer la commande, cliquer sur la flèche verte du côté droit du chunk. Le résultat apparaît alors dessous et la commande tourne dans la console.

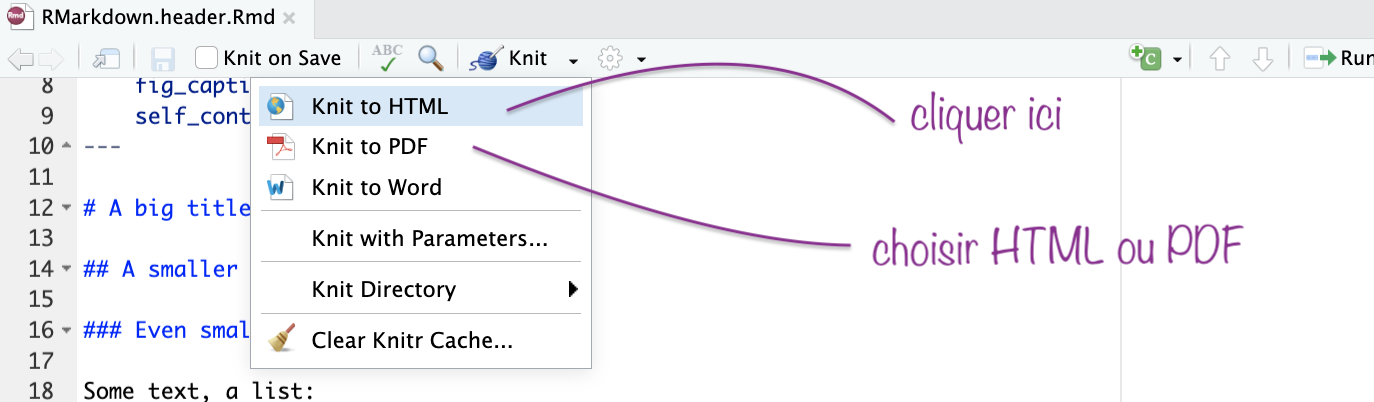
Produire un rapport (PDF ou HTML)
Cliquer sur le bouton knit et choisir le format.
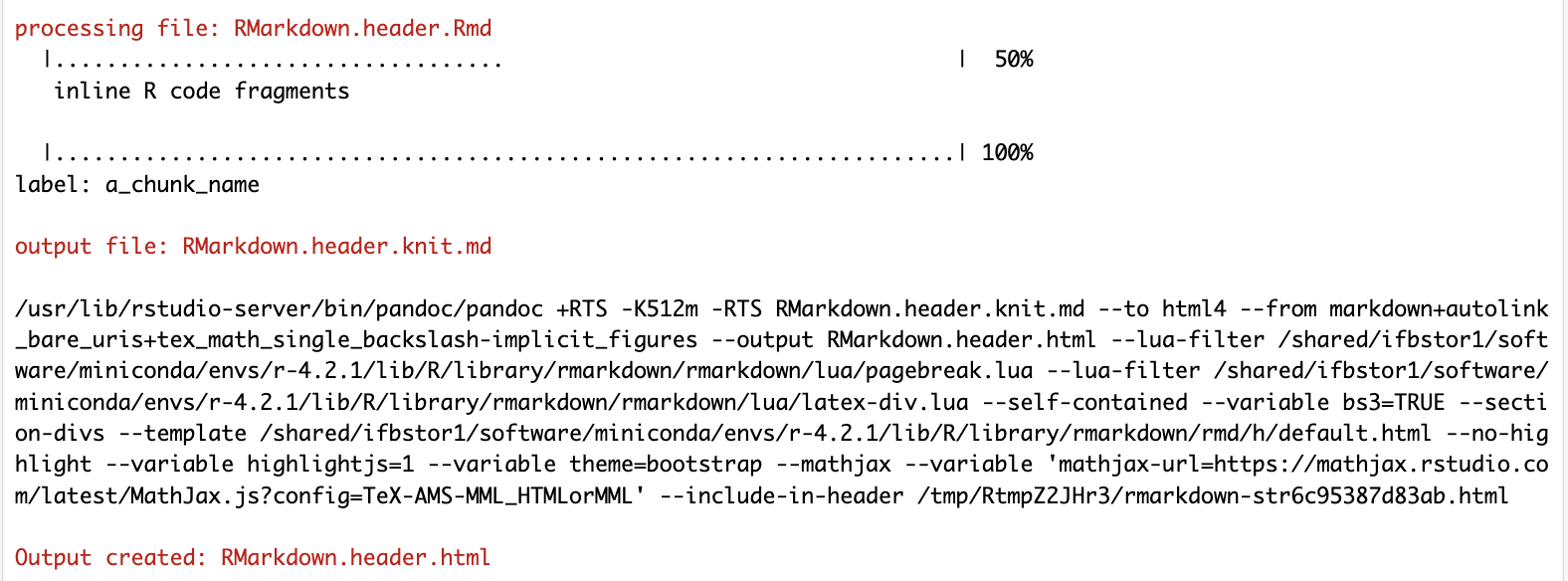
L'ensemble des commandes va être lancé. Attention, cela peut prendre du temps ! Les étapes apparaissent dans la console, le fichier de sortie (ici un rapport au format html) est indiqué. Il se trouve dans le même dossier que le fichier R markdown.
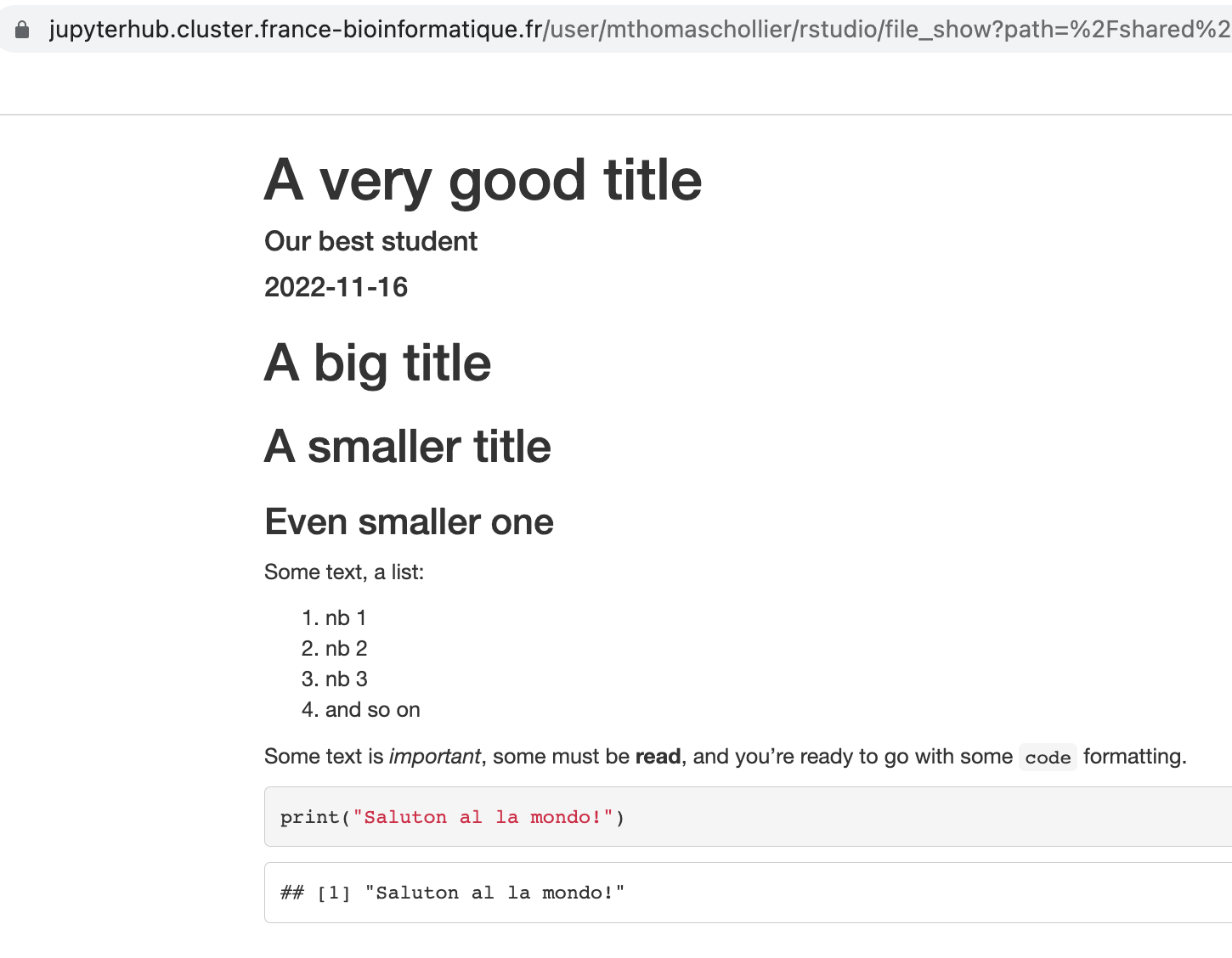
Vous pouvez ouvrir ce rapport dans votre navigateur web. Pour chaque chunk, la commande est indiquée ainsi que son résultat, y compris les figures.
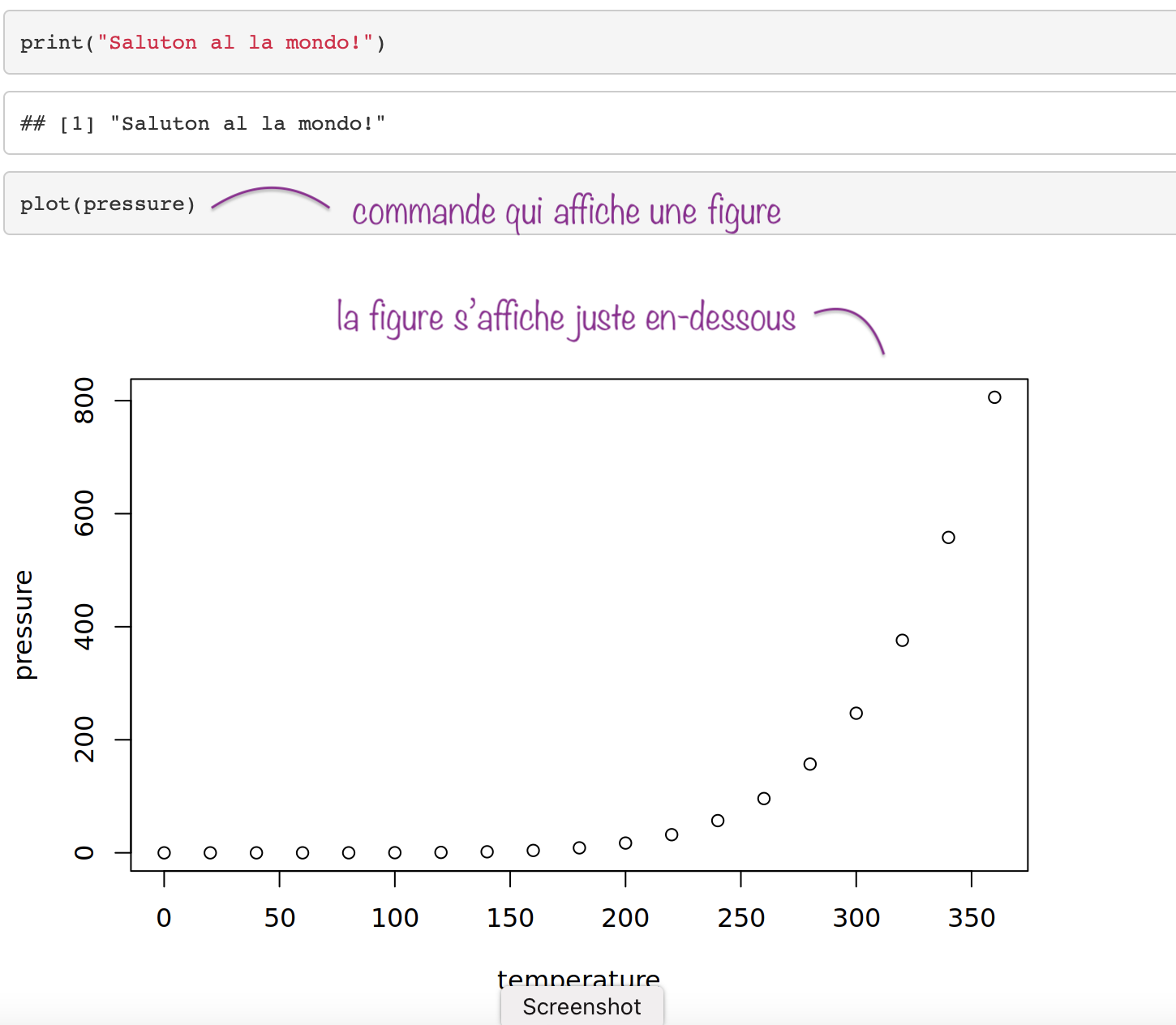
Par exemple, vous pouvez ajouter le chunk suivant pour afficher des données d'exemple déjà incluses dans R. Ici, nous avons choisi les données nommées pressure.
Après avoir généré le rapport avec le bouton knit, le fichier html comporte la commande et la figure.